Erläuterung der Schätzung der zeitlich variierenden Reproduktionszahl R
Dieses Dokument ist Teil der Anfrage „Verfahren zur R-Zahl Berechnung“
Erläuterung der Schätzung der zeitlich variierenden Reproduktionszahl R Robert Koch-Institut 15. Mai 2020 Zusammenfassung Die Berechnung des 7-Tages R-Werts wird methodisch und anhand einer Implementation in der Statistik-Software R erläutert. Dieses Dokument richtet sich an das epidemiologische Fachpublikum. Hintergrund In an der Heiden und Hamouda (2020) wurde das Verfahren des RKI zur Bestimmung der zeitlich variierenden Reproduktionszahl, des sogenannten R-Werts, beschrieben. Das Verfahren besteht aus drei Schritten: 1. Multiple Imputation fehlender Information zum Erkrankungsbeginn von COVID-19- Fällen unter einer Missing-at-Random Annahme 2. Korrektur der Anzahl von Neuerkrankungen für den Diagnose-, Melde- und Übermittlungsverzug mittels des Nowcasting-Verfahren 3. Berechnung der zeitlich variierenden Reproduktionszahl unter der Annahme einer Generationszeit von 4 Tagen Die Schritte 1 und 2 führen zu einer geschätzten epidemischen Kurve, welche Einschätzungen zum Trend und Umfang des Ausbruchs anhand von absoluten Fallzahlen erlaubt. Schritt 3, die Berechnung des zeitlich variierenden R-Werts, entspricht einer Trendanalyse dieser epidemischen Kurve. Der R-Wert ist eine epidemiologische Kennzahl, um die Dynamik des Ausbruchsgeschehens zu beschreiben. In dem vorliegenden Dokument soll auf die R-Wert Bestimmung (Berechnungen in Schritt 3) genauer eingegangen werden. Speziell soll die Berechnung des sogenannten 7-Tages R- Werts mathematisch erläutert werden. Dieser unterscheidet sich von dem bereits berichteten sensitiveren R-Wert durch eine erweiterte Glättung, die die statistische Schätzunsicherheit verringert. Somit ist der 7-Tages R-Wert in seiner zeitlichen Dynamik stabiler und reagiert weniger sensitiv auf die momentane Einschätzung der epidemischen Kurve durch das Nowcasting. Erläuterung der R-Schätzung Mit Hilfe der vom RKI bereitgestellten Excel-Tabelle der aktuellen Schätzung durch Imputation und Nowcast lässt sich die R-Wert Berechnung des Schritt 3 des Verfahrens 1

nachrechnen und visualisieren. Siehe hierzu https://www.rki.de/DE/Content/InfAZ/N/Neuartiges_Coronavirus/Projekte_RKI/Nowcast ing.html Das RKI verwendet zur Schätzung der zeitlich variierenden Reproduktionszahl R aufgrund des geschätzten Verlaufs der Anzahl von Neuerkrankungen 𝐸𝐸𝑡𝑡 die folgende Formel nach Cori et al. (2013): 𝐸𝐸𝑡𝑡 𝑅𝑅𝑡𝑡 = , 𝛬𝛬𝑡𝑡 𝑡𝑡 ∑𝑠𝑠=1 𝐸𝐸𝑡𝑡−𝑠𝑠 wobei 𝛬𝛬𝑡𝑡 = 𝑤𝑤𝑠𝑠 und 𝑤𝑤1 , 𝑤𝑤2 , … die diskrete Wahrscheinlichkeits-Verteilung des seriellen Intervalls mit Träger 1,2, … bezeichnet, d.h. für 𝑖𝑖 = 1,2, … gilt 0 ≤ 𝑤𝑤𝑖𝑖 ≤ 1 und die Summe über alle 𝑤𝑤𝑖𝑖 ist 1. In der Formel wird also angenommen, dass die neuen Erkrankungsfälle 𝐸𝐸𝑡𝑡 zum Zeitpunkt 𝑡𝑡 sich jeweils bei einem Anteil 𝑤𝑤𝑡𝑡 der früher erkrankten Personen 𝐸𝐸𝑡𝑡−𝑠𝑠 angesteckt haben. Rein technisch handelt es sich bei 𝑅𝑅𝑡𝑡 um eine sog. instantaneous reproduction number [Cori et al. (2013)], welche rückwärts-schauend in der Zeit definiert ist. Unter der Annahme einer konstanten Generationszeit und eines konstanten seriellen Intervalls von 4 Tagen ergibt sich daraus zunächst die Formel 𝐸𝐸𝑡𝑡 𝑅𝑅𝑡𝑡 = , 𝐸𝐸𝑡𝑡−4 weil bei dieser Annahme die Verteilung des seriellen Intervalls gleich 𝑤𝑤𝑖𝑖 ≡ 𝐼𝐼(𝑖𝑖 = 4) ist, wobei 𝐼𝐼() die Indikatorfunktion angibt. Das heißt 𝑅𝑅𝑡𝑡 gibt an, wieviele Personen eine Person mit Erkrankungsbeginn zum Zeitpunkt 𝑡𝑡 − 4 im Durchschnitt ansteckt. Die angesteckten Personen werden dann zum Zeitpunkt 𝑡𝑡 beobachtet. Die obige Schätzung von R verhält sich allerdings typischerweise relativ unruhig und wird normalerweise nicht verwendet - vgl. z.B. Cori et al. (2013), S. 1506. Statt 𝑅𝑅𝑡𝑡 nur für einen Zeitpunkt 𝑡𝑡 zu berechnen, kann 𝑅𝑅𝑡𝑡 auch über ein Intervall von 𝜏𝜏 Tagen berechnet werden. Cori et al. zeigen, dass dafür die folgende Formel genutzt werden kann: 𝑡𝑡 ∑𝑠𝑠=𝑡𝑡−𝜏𝜏+1 𝐸𝐸𝑠𝑠 𝑅𝑅𝑡𝑡,𝜏𝜏 = 𝑡𝑡 , ∑𝑠𝑠=𝑡𝑡−𝜏𝜏+1 𝛬𝛬𝑠𝑠 Beträgt das serielle Intervall 4 Tage, dann vereinfacht sich diese Formel zu 𝑡𝑡 ∑𝑠𝑠=𝑡𝑡−𝜏𝜏+1 𝐸𝐸𝑠𝑠 𝑅𝑅𝑡𝑡,𝜏𝜏 = 𝑡𝑡 . ∑𝑠𝑠=𝑡𝑡−𝜏𝜏+1 𝐸𝐸𝑠𝑠−4 Diese Formel kann äquivalent auch als Quotient zweier gleitendender Mittel über 𝜏𝜏 Tage der 𝐸𝐸𝑠𝑠 -Werte beschrieben werden, also als 1 𝑡𝑡 𝜏𝜏 ∑𝑠𝑠=𝑡𝑡−𝜏𝜏+1 𝐸𝐸𝑠𝑠 𝐸𝐸𝑡𝑡 𝑅𝑅𝑡𝑡,𝜏𝜏 = 𝜏𝜏 ≡ 𝜏𝜏 , 1 𝑡𝑡 ∑𝑠𝑠=𝑡𝑡−𝜏𝜏+1 𝐸𝐸𝑠𝑠−4 𝐸𝐸𝑡𝑡−4 𝜏𝜏 2

𝜏𝜏 1 𝑡𝑡 ∑𝑠𝑠=𝑡𝑡−𝜏𝜏+1 𝐸𝐸𝑠𝑠 wobei 𝐸𝐸𝑡𝑡 = den gleitende Mittelwert der Anzahl von Neuerkrankungen über 𝜏𝜏 𝜏𝜏-Tage bezeichnet. Der bisherige vom RKI berechnete (sensitive) R-Wert ergibt sich für 𝜏𝜏 = 4, also als 4 𝑡𝑡 ∑𝑠𝑠=𝑡𝑡−3 𝐸𝐸𝑠𝑠 𝐸𝐸𝑡𝑡 𝑅𝑅𝑡𝑡,4 = 4 = 𝑡𝑡 . 𝐸𝐸𝑡𝑡−4 ∑𝑠𝑠=𝑡𝑡−3 𝐸𝐸𝑠𝑠−4 Der stabilere 7-Tages R-Wert ergibt sich für ein Glättungsintervall von 𝜏𝜏 = 7 Tagen, also als 7 𝑡𝑡 ∑𝑠𝑠=𝑡𝑡−6 𝐸𝐸𝑠𝑠 𝐸𝐸𝑡𝑡 𝑅𝑅𝑡𝑡,7 = 7 = 𝑡𝑡 . 𝐸𝐸𝑡𝑡−4 ∑𝑠𝑠=𝑡𝑡−6 𝐸𝐸𝑠𝑠−4 Der an Tag 𝑢𝑢 berichtete R-Wert bezieht sich auf das Nowcasting bis zum Zeitpunkt 𝑡𝑡 = 𝑢𝑢 − 4 und damit in der sensitiven Variante auf Neuerkrankungen im Zeitraum 𝑢𝑢 − 7, ⋯ , 𝑢𝑢 − 4 und in der stabileren Variante auf Neuerkrankungen im Zeitraum 𝑢𝑢 − 10, ⋯ , 𝑢𝑢 − 4. Beide Variantes des R-Wertes beziehen sich also auf Intervalle und werden nur zu Darstellungszwecken einem einzelnen Tag zugeordnet. Bezieht man noch die Inkubationszeit von 4 bis 6 Tagen mit ein, so beschreibt die am Tag 𝑢𝑢 berichtete Reproduktionszahl 𝑅𝑅𝑡𝑡 in der sensitiven Variante die Neuinfektionen im Zeitraum 𝑢𝑢 − 13, ⋯ , 𝑢𝑢 − 8 und in der stabileren Variante die Neuinfektionen im Zeitraum 𝑢𝑢 − 16, ⋯ , 𝑢𝑢 − 8. Dieses letztere Intervall reicht im Vergleich länger zurück und lässt sich eher mit dem Intervall 𝑢𝑢 − 14, ⋯ , 𝑢𝑢 − 9 als mit dem Intervall 𝑢𝑢 − 13, ⋯ , 𝑢𝑢 − 8 vergleichen. Um den R-Wert und den 7-Tage R-Wert besser vergleichen zu können, wird daher der 7- Tage R-Wert um einen Tag zurück datiert. Siehe dazu auch Abbildung 2. Als Beispiel: Im RKI-Lagebericht am 15. Mai 2020 bezieht sich der angegebene sensitive R- Wert auf das Infektionsgeschehen im Zeitraum vom 02. Mai 2020 bis 07. Mai 2020. Der stabile R-Wert auf den Zeitraum 29. April 2020 bis 07. Mai 2020. Die 95% Prädiktionsintervalle für diese beiden R-Werte für einen spezifischen Tag 𝑡𝑡 ergeben sich durch Anwendung der obigen Formel für die R-Werte der 200 Realisationen des Nowcastings. Sie können nicht auf einfache Weise aus den in der Excel-Tabelle angegeben Prädiktionsintervallen für die Neuerkrankungen erzeugt werden. Wichtig ist, dass es sich bei beiden R-Werten um eine statistische Schätzung handelt, weshalb die Prädiktionsintervalle wichtige Informationen zur Sicherheit der Schätzung enthalten. Implementation in R # Lade neuesten Nowcast von der RKI Webseite daten_file <- str_c("Nowcasting_Zahlen-",Sys.Date(),".xlsx") if (!file.exists(daten_file)) { file_url <- "https://www.rki.de/DE/Content/InfAZ/N/Neuartiges_Coronavirus/Projekte_RKI/No wcasting_Zahlen.xlsx?__blob=publicationFile" download.file(url=file_url,destfile= daten_file, mode="wb") } 3

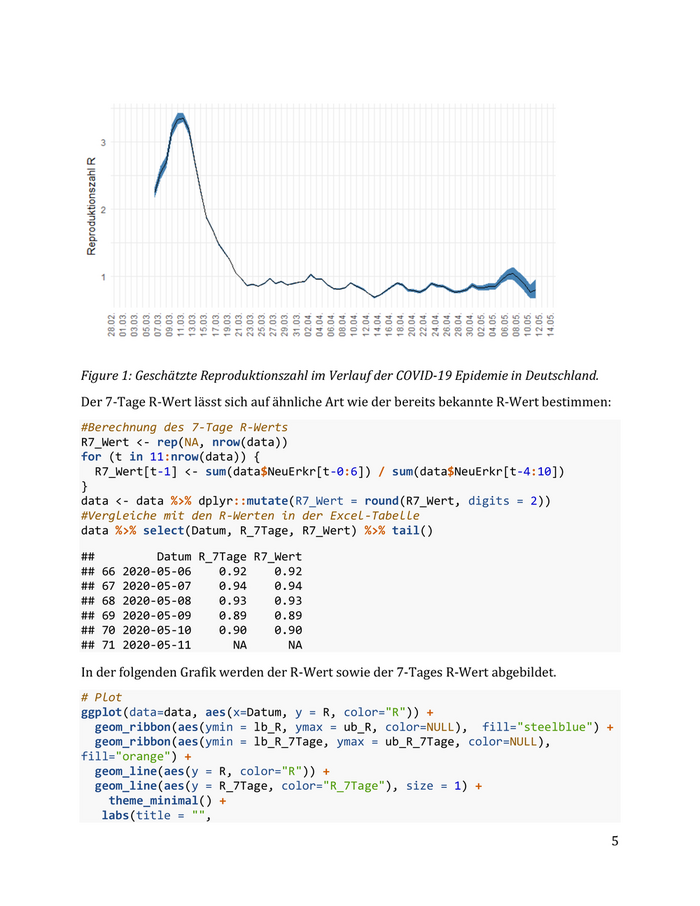
# Lese Excel-File data <- xlsx::read.xlsx(file = daten_file, sheetName = "Nowcast_R", encoding = "UTF-8") data <- data[,1:13] # Umbennung der Spalten Namen zu kürzeren Variabelnamen names(data) <- c("Datum", "NeuErkr", "lb_NeuErkr", "ub_NeuErkr", "NeuErkr_ma4", "lb_NeuErkr_ma4", "ub_NeuErkr_ma4", "R", "lb_R", "ub_R", "R_7Tage", "lb_R_7Tage", "ub_R_7Tage") # R-Wert Berechnung bei einem seriellen Intervall von 4 Tagen R_Wert <- rep(NA, nrow(data)) for (t in 8:nrow(data)) { R_Wert[t] <- sum(data$NeuErkr[t-0:3]) / sum(data$NeuErkr[t-4:7]) } data <- data %>% dplyr::mutate(R_Wert = round(R_Wert, digits = 2)) #Vergleiche mit den R-Werten in der Excel-Tabelle data %>% select(Datum, R, R_Wert) %>% tail() ## Datum R R_Wert ## 66 2020-05-06 1.02 1.02 ## 67 2020-05-07 1.04 1.04 ## 68 2020-05-08 0.97 0.97 ## 69 2020-05-09 0.88 0.88 ## 70 2020-05-10 0.77 0.77 ## 71 2020-05-11 0.80 0.80 Unterschiede in der dritten Nachkommastelle des nachgerechneten R-Wertes entstehen durch etwas unterschiedliche Verwendung von Rundungen auf ganze Zahlen. # Plot ggplot(data=data, aes(x=Datum)) + geom_ribbon(aes(ymin = lb_R, ymax = ub_R), stat="identity", fill="steelblue")+ geom_line(aes(y = R), stat="identity", fill="steelblue")+ theme_minimal() + labs(title = "", x = "", y = "Reproduktionszahl R") + scale_x_date(date_breaks = "2 days", labels = scales::date_format("%d.%m.")) + scale_y_continuous(labels = function(x) format(x, big.mark = ".", decimal.mark = ",", scientific = FALSE)) + theme(axis.text.x = element_text(angle=90, vjust=0)) 4

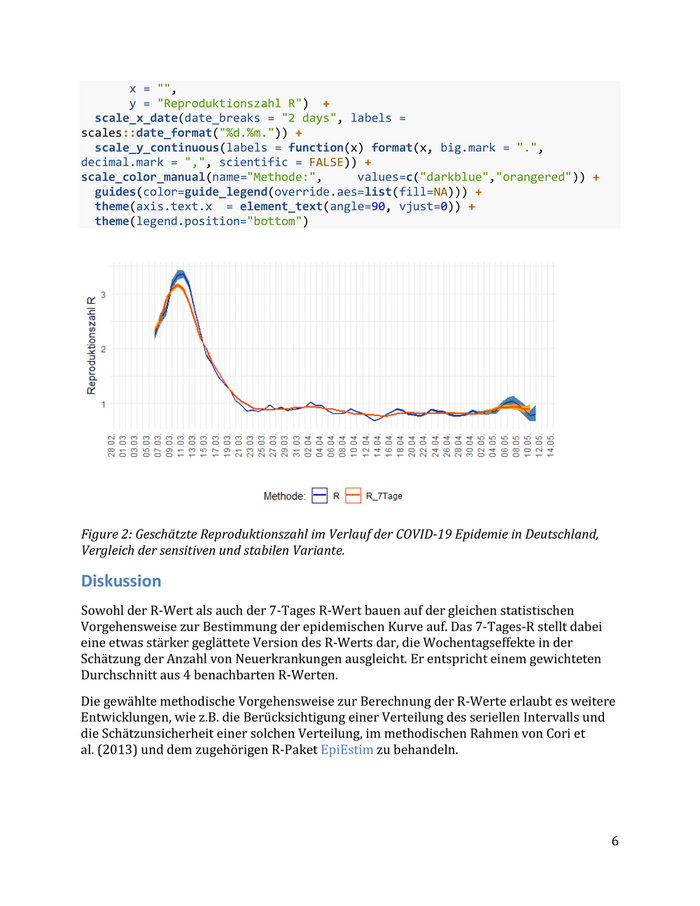
Figure 1: Geschätzte Reproduktionszahl im Verlauf der COVID-19 Epidemie in Deutschland. Der 7-Tage R-Wert lässt sich auf ähnliche Art wie der bereits bekannte R-Wert bestimmen: #Berechnung des 7-Tage R-Werts R7_Wert <- rep(NA, nrow(data)) for (t in 11:nrow(data)) { R7_Wert[t-1] <- sum(data$NeuErkr[t-0:6]) / sum(data$NeuErkr[t-4:10]) } data <- data %>% dplyr::mutate(R7_Wert = round(R7_Wert, digits = 2)) #Vergleiche mit den R-Werten in der Excel-Tabelle data %>% select(Datum, R_7Tage, R7_Wert) %>% tail() ## Datum R_7Tage R7_Wert ## 66 2020-05-06 0.92 0.92 ## 67 2020-05-07 0.94 0.94 ## 68 2020-05-08 0.93 0.93 ## 69 2020-05-09 0.89 0.89 ## 70 2020-05-10 0.90 0.90 ## 71 2020-05-11 NA NA In der folgenden Grafik werden der R-Wert sowie der 7-Tages R-Wert abgebildet. # Plot ggplot(data=data, aes(x=Datum, y = R, color="R")) + geom_ribbon(aes(ymin = lb_R, ymax = ub_R, color=NULL), fill="steelblue") + geom_ribbon(aes(ymin = lb_R_7Tage, ymax = ub_R_7Tage, color=NULL), fill="orange") + geom_line(aes(y = R, color="R")) + geom_line(aes(y = R_7Tage, color="R_7Tage"), size = 1) + theme_minimal() + labs(title = "", 5
x = "", y = "Reproduktionszahl R") + scale_x_date(date_breaks = "2 days", labels = scales::date_format("%d.%m.")) + scale_y_continuous(labels = function(x) format(x, big.mark = ".", decimal.mark = ",", scientific = FALSE)) + scale_color_manual(name="Methode:", values=c("darkblue","orangered")) + guides(color=guide_legend(override.aes=list(fill=NA))) + theme(axis.text.x = element_text(angle=90, vjust=0)) + theme(legend.position="bottom") Figure 2: Geschätzte Reproduktionszahl im Verlauf der COVID-19 Epidemie in Deutschland, Vergleich der sensitiven und stabilen Variante. Diskussion Sowohl der R-Wert als auch der 7-Tages R-Wert bauen auf der gleichen statistischen Vorgehensweise zur Bestimmung der epidemischen Kurve auf. Das 7-Tages-R stellt dabei eine etwas stärker geglättete Version des R-Werts dar, die Wochentagseffekte in der Schätzung der Anzahl von Neuerkrankungen ausgleicht. Er entspricht einem gewichteten Durchschnitt aus 4 benachbarten R-Werten. Die gewählte methodische Vorgehensweise zur Berechnung der R-Werte erlaubt es weitere Entwicklungen, wie z.B. die Berücksichtigung einer Verteilung des seriellen Intervalls und die Schätzunsicherheit einer solchen Verteilung, im methodischen Rahmen von Cori et al. (2013) und dem zugehörigen R-Paket EpiEstim zu behandeln. 6
Literatur • an der Heiden, M, Hamouda, O, “Schätzung der aktuellen Entwicklung der SARS-CoV-2- Epidemie in Deutschland - Nowcasting”, Epid Bull 2020;17:10–16, https://doi.org/10.25646/6692.4 • Cori A, Ferguson NM, Fraser C, and Cauchemez S, “A New Framework and Software to Estimate Time-Varying Reproduction Numbers During Epidemics”, American journal of epidemiology 178(9), 1505-1512, https://doi.org/10.1093/aje/kwt133 7







